Introducción
Los coronavirus infectan a la población humana causando enfermedades respiratorias moderadas, pero con frecuencia se presentan casos con mayor gravedad que incluso pueden llegar a ser fatales. El SARS-CoV-2 es uno de los siete virus de la familia Coronaviridae que infectan a humanos, cuatro de los cuales (229E, OC43, NL63 y HKU1) causan enfermedades estacionales con síntomas similares a los del resfriado común1,2. Otros virus de la familia recientemente descubiertos son el que causa el síndrome respiratorio agudo severo (SARS-CoV) y el coronavirus que causa el síndrome respiratorio del Medio Oriente (MERS-CoV), que son de origen zoonótico y se han relacionado con enfermedades agudas y mortales, comprometiendo de forma grave al sistema respiratorio y, por lo tanto, la vida de los hospederos3.
Los coronavirus han provocado brotes o pandemias, como la ocasionada en 2002-2003 por el SARS-CoV, infectando a 8437 personas con una mortalidad del 9.6%4. En 2012, el MERS-CoV infectó a 2494 personas, presentando una mortalidad del 34.4%5. Finalmente, la reciente pandemia en 2019-2020 por el SARS- CoV-2 provocó la enfermedad denominada COVID-19 (Coronavirus Disease 2019), y hasta el momento se han documentado 700 millones de casos confirmados y aproximadamente 7 millones de decesos6. La pandemia de COVID-19 inició en diciembre de 2019 en la ciudad de Wuhan, provincia de Hubei, China, en un grupo de personas que presentaron una neumonía de origen desconocido, con sospecha de haberse contagiado en un mercado donde se vendían alimentos de origen animal7.
Biología del SARS-CoV-2
Los coronavirus son virus envueltos con genoma de RNA con polaridad positiva. El tamaño del genoma es de 30 a 32 kb y pertenecen al orden Nidovirales, familia Coronaviridae, subfamilia Coronavirinae2. Dentro de esta subfamilia se encuentran cuatro diferentes géneros: Alfacoronavirus, Betacoronavirus, Gammacoronavirus y Deltacoronavirus; el SARS-CoV-2 pertenece a los Betacoronavirus7. La proteína de la nucleocápside forma complejos con el RNA del genoma para configurar una estructura helicoidal de la cápside que se encuentra dentro de la envoltura viral. La proteína espícula o Spike (S) se encuentra en forma de trímeros, los cuales forman unas estructuras denominadas peplómeros que están embebidas en la envoltura del virión, dándole la forma de una corona. En algunos viriones de coronavirus, la proteína de envoltura (E) forma picos más pequeños en la membrana8 (Fig. 1).
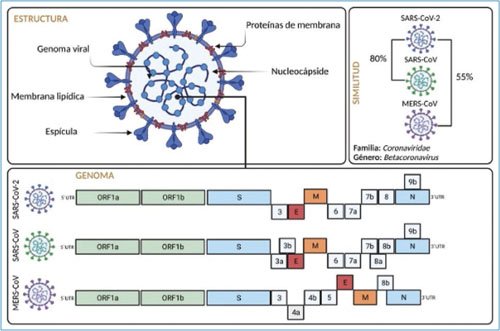
Figura 1. Biología viral: estructura, proteínas y genoma del SARS-CoV-2, agente etiológico de la COVID-19. Comparación de la estructura del genoma con otros betacoronavirus humanos más cercanos (SARS-CoV y MERS-CoV), y homología de nucleótidos.
El extremo 5’ del genoma del coronavirus codifica el gen de la replicasa, el cual contiene dos marcos de lectura abiertos (ORF, open reading frame), ORF1a y ORF1ab, que abarcan 20 kb, aproximadamente dos tercios del genoma (Fig. 1). Los ORF 1a y 1ab codifican para 16 proteínas no estructurales (NSP, non-structural protein). Muchas de estas NSP tienen actividades enzimáticas, incluidas proteasas, enzimas de modificación de RNA, así como polimerasas y helicasas. En el extremo 3’ se encuentra la región que codifica para las proteínas estructurales: membrana (M), envoltura (E), nucleocápside (N) y Spike (S). Además, en el extremo 3’ se encuentra también la región que codifica para las proteínas accesorias 3, 6, 7a, 7b, 8 y 9b, de las cuales aún no se conoce en profundidad su función, aunque se sabe que pueden tener funciones de inhibición de la respuesta inmunitaria innata9.
Ciclo de replicación
La infección a la célula blanco comienza con la unión al receptor de la enzima convertidora de angiotensina 2 (ACE2, angiotensin-converting enzyme 2) y el corte proteolítico de la proteína S, ya sea de forma extracelular por la serina proteasa transmembrana-2 o por la furina, en vesículas citoplásmicas (Fig. 2). El corte proteolítico en la proteína S da como resultado la formación de dos subunidades S1 y S2; en esta última se encuentra el péptido fusión que interacciona con la membrana celular dando como resultado final la introducción del RNA genómico viral al citoplasma10. Debido a que el RNA genómico es de polaridad positiva, este puede ser tomado como molde por los ribosomas celulares para la producción de los polipétidos ORF1a y ORF1ab. Las primeras proteínas que se sintetizan a partir de estos polipéptidos forman un complejo de replicación y son principalmente enzimas (RNA polimerasa dependiente de RNA) encargadas de la transcripción y la producción de RNA subgenómicos y genómicos. Los RNA subgenómicos codifican para las proteínas estructurales (N, E, M y S) y para las proteínas accesorias. El RNA genómico es ensamblado con las proteínas de nucleocápside y las proteínas virales ancladas en membranas celulares, formando nuevos viriones que son liberados de la célula por medio de gemación11 (Fig. 2).
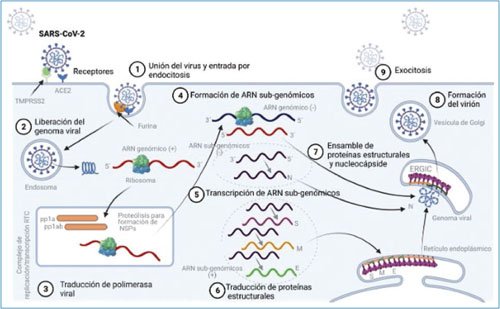
Figura 2. Ciclo de replicación del SARS-CoV-2. Reconocimiento del receptor celular, entrada y fusión de las membranas celular y viral para el posterior ingreso del genoma viral al citoplasma. El ciclo se completa con la transcripción viral y la síntesis de proteínas para el ensamble de nuevas partículas virales y su liberación por gemación. ERGIC: compartimento intermedio retículo endoplasmático-Golgi; NSP: proteínas no estructurales.
Origen
Hasta el momento no existen evidencias suficientes que indiquen cuándo y dónde surgió este nuevo virus, pero la teoría más aceptada es la de la transmisión zoónotica de murciélago a un hospedero intermediario y finalmente al humano. Esta hipótesis está fundamentada principalmente en el análisis de las secuencias nucleotídicas de otros Betacoronavirus presentes en algunos mamíferos, como roedores y murciélagos7. Los Betacoronavirus con mayor homología de nucleótidos son los encontrados en murciélagos de la especie Rhinolophus affinis y Rhinolophus sinicus, los cuales presentan una homología del 96% y el 86%, respectivamente. Otro Betacoronavirus muy similar en secuencia es el presente en el mamífero Manis javanica (pangolín), el cual tiene una homología con el SARS-CoV-2 del 91%; sin embargo, presenta una homología de más del 95% (Fig. 3) en la región del dominio de unión al receptor (RBD, receptor-binding domain)12. Estas tres especies podrían estar en contacto frecuente debido al tráfico ilegal de especies vivas para su posterior consumo humano en China. Su interacción podría ser el mecanismo por el cual los virus de murciélagos infectan a otros mamíferos que puedan funcionar como hospederos intermediarios para finalmente infectar a los humanos.
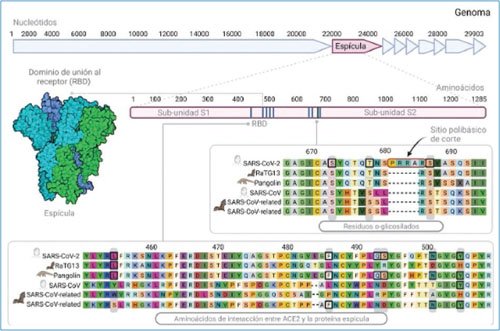
Figura 3. Proteína Spike (S) del SARS-CoV-2 y dominio de unión al receptor (RBD). La proteína S tiene aproximadamente 1300 aminoácidos y en la región S1 se encuentra el RBD; se conocen al menos seis posiciones de interacción con el receptor celular ACE2. En la región S2 se encuentra el sitio polibásico de corte (recuadros). Se muestra una alineación de la secuencia de aminoácidos de estas regiones comparando la secuencia del SARS-CoV-2 con la de otros betacoronavirus de aves y otros mamíferos. ACE2: enzima convertidora de la angiotensina 2.
La proteína Spike del SARS-CoV-2
La proteína S es el mayor antígeno de superficie del virus y su función principal es la de unirse con el receptor celular y mediar la fusión de membranas requerida para la entrada a la célula y el inicio de la infección; por lo tanto, determina el rango de los hospederos y el tropismo viral13. Debido a lo anterior, la proteína S es el blanco principal de los anticuerpos neutralizantes generados durante la infección, siendo también el objetivo en el diseño de vacunas.
La proteína S es una proteína trimérica y cada monó-mero se sintetiza como una única cadena polipeptídica de aproximadamente 1300 aminoácidos. En muchos coronavirus, la proteína S es procesada por proteasas del huésped generando dos subunidades funcionales (S1 y S2) (Fig. 3) que permanecen unidas de forma no covalente en la conformación prefusión11. Estas estructuras son importantes para el reconocimiento del receptor celular, la entrada y la fusión del virus10. En la subunidad S1 se encuentra la región RBD que incluye los aminoácidos 357 a 528. Dentro de esta región existen al menos seis posiciones que son esenciales para la unión con el receptor celular: 445, 486, 493, 494, 501 y 505 (Fig. 3). Adyacentes a estas posiciones se encuentran aminoácidos que son importantes para la presentación de antígenos y la estimulación de la respuesta inmunitaria humoral y celular13.
Receptor celular ACE2 y tropismo viral
La proteína ACE2 es miembro de una familia que tiene amplia distribución en varios tejidos. En cuanto a su función, ACE2 presenta diversas funciones biológicas, entre ellas la regulación de la presión sanguínea a través del sistema renina angiotensina-aldosterona, que se vincula con la patogénesis de enfermedades cardiovasculares14. En particular, en el páncreas tiene un papel importante como protector de la glucemia, mientras que en el riñón unos niveles bajos de ACE2 se asocian con enfermedades progresivas como la nefropatía diabética14,15.
Una de las características principales de la COVID-19 es la gran cantidad de órganos que se ven afectados, en gran parte debido a que muchos órganos expresan la proteína ACE2. Los datos histopatológicos han mostrado que el virus tiene tropismo por diferentes estirpes celulares y órganos, como los sistemas renal, circulatorio y neurológico, así como por el tejido faríngeo y gastrointestinal, y de manera principal por el tracto respiratorio16. Es importante mencionar que después de iniciada la infección por SARS-CoV-2 se generan efectos secundarios en la fisiopatología de varios órganos, así como toxicidad viral directa, dañando a las células endoteliales, provocando tromboinflamación y desregulación del sistema renina angiotensina-aldosterona. Cabe señalar que se cree que la alteración en el sistema renina angiotensina-aldosterona puede ser exclusiva de la COVID-1916. Asimismo, la patogenia inmunitaria causada por la hiperproducción y la liberación de citocinas proinflamatorias que afectan de manera importante a los pulmones, así como la desregulación en la microcirculación, podrían ser también consecuencia de la sepsis viral17.
La infección por SARS-CoV-2 en humanos es posible por la existencia de receptores celulares específicos del hospedero (SARS-CoV, SARS-CoV-2 y HCoV-NL63 reconocen a ACE2, MERS-CoV reconoce a una dipeptidil peptidasa 4, y OC43 y HKU1 reconocen a 5-N- acetil-9-O-acetil-sialidasa) que permiten la entrada del virus a la célula, para posteriormente completar su ciclo y dispersarse de manera efectiva en la población humana18. Por otro lado, se ha descrito que los polimorfismos de ACE2 en células humanas podrían modificar la interacción con SARS-CoV-2, aumentando o disminuyendo la afinidad de ACE2 con el RBD de la proteína S del virus y favoreciendo, en algunos casos, la infección viral19.
Evolución natural de la enfermedad
La infección comienza con la exposición directa e indirecta a una fuente de contagio, lo cual permite la entrada del virus al sistema respiratorio y la interacción de este con el receptor ACE2 presente en las células epiteliales20 (Fig. 4). Dado que existe una amplia distribución del receptor ACE2 en las vías respiratorias altas, el establecimiento de la infección es muy probable, asociándose con una alta carga viral en el momento de la exposición21. Los principales síntomas respiratorios comienzan a manifestarse entre 5 y 7 días después de la exposición. A partir del establecimiento de la infección en el tracto respiratorio alto y la cavidad orofaríngea, se disemina a diferentes órganos y se convierte en una infección sistémica. Algunas células no epiteliales de la mucosa, la tráquea y los bronquios presentan también el receptor ACE2, incluyendo a las células alveolares tipo II que se encuentran en el parénquima pulmonar, indicando que este receptor está presente no solo en células epiteliales, sino también en varias partes del tracto respiratorio. Por ello, el epitelio traqueal y bronquial también es susceptible a la infección por SARS-CoV-2. Cabe destacar que estos sitios primarios de infección podrían facilitar la propagación del virus a los alvéolos, donde ocurre el daño principal y donde la expresión de ACE2 puede ser estimulada por procesos ambientales, como infecciones microbianas o procesos inflamatorios en el pulmón20. La primera respuesta inmunitaria contra el virus es la respuesta innata, compuesta principalmente por interferones, y comienza antes o durante el inicio de los síntomas (Fig. 4). Esta respuesta es primordial para una restricción de la replicación viral en estado temprano y para delimitar la infección a sus formas menos graves22. Posteriormente aparece la respuesta inmunitaria adaptativa, compuesta principalmente de linfocitos B, y la producción de anticuerpos neutralizantes y de linfocitos T con la respuesta celular23. La respuesta de anticuerpos neutralizantes parece durar más de 1 año y su magnitud depende sobre todo de la exposición a los antígenos virales, siendo los pacientes con enfermedad grave los que tienen mayor cantidad de estos anticuerpos comparados con los pacientes que cursan asintomáticos o con síntomas moderados24.
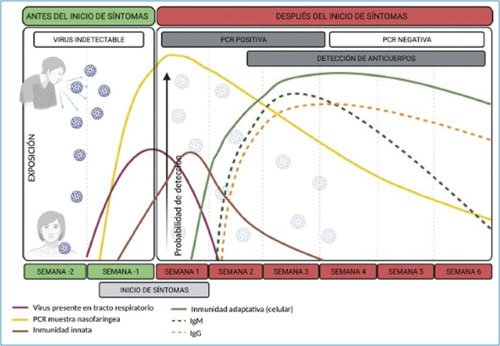
Figura 4. Evolución natural de la infección por SARS-CoV-2. Después de la exposición al virus se establece la infección en las vías respiratorias altas, que inicia con síntomas respiratorios entre 5 y 7 días tras la exposición. El virus puede detectarse antes del inicio de los síntomas en la cavidad nasofaríngea y orofaríngea, y sigue excretándose al menos 3 semanas más. Las respuestas inmunitarias contra el virus de tipo innata, de anticuerpos y celular aparecen posterior al inicio de los síntomas. IgG: inmunoglobulina G; IgM: inmunoglobulina M; PCR: reacción en cadena de la polimerasa.
¿Ha cambiado el SARS-CoV-2 desde su aparición en Wuhan?
Debido a su naturaleza como virus de genoma de RNA, el SARS-CoV-2 presenta una alta tasa de mutación25, aunque esta es menor en comparación con otros virus de RNA, como el de la influenza26. Varios factores están implicados en esta variación biológica: una alta transmisión en la población, factores del hospedero y el uso de vacunas pueden aumentarla, y por lo tanto favorecer la aparición de cambios o mutaciones a lo largo del genoma viral27. Desde su aparición a finales de 2019, la mayoría de las proteínas han sufrido cambios, principalmente NSP3, NSP12b, ORF3a, N, NSP2, NSP4, ORF8, NSP14, NSP13 y, de manera importante, la proteína S28.
El primer cambio de relevancia fue en el sitio 614 (D614G), el cual fue detectado inicialmente en Inglaterra y se expandió con rapidez por Europa y Norteamérica. Este cambio definió la aparición de nuevos clados del virus (G, GH, GV, GR, GK), que predominaron en Europa y Norteamérica a partir de marzo de 2020. Los estudios in vitro y epidemiológicos indican que este cambio confirió al virus una mayor capacidad de transmisión y más patogenicidad29. El cambio en la posición 614 recae en la zona de interacción de los monómeros de la proteína S, aumentando la habilidad para adoptar una conformación abierta, que es la que tiene mayor afinidad con el receptor celular30. A partir de septiembre de 2020 comenzaron a detectarse en diferentes partes del mundo otros cambios en la proteína S, como N501Y, E484K, K417N, L452R y P681H28,30. Estas mutaciones se encuentran en regiones como la RDB, en las interfaces monómero-monómero y en el sitio de corte por la furina celular, así como en sitios inmunogénicos30. Es importante realizar una vigilancia molecular para detectar estas mutaciones en los virus SARS-CoV-2 de forma local, regional y mundial.
Una variante viral presenta dos o más mutaciones que pueden conferir un fenotipo de mayor transmisión, de evasión de la respuesta inmunitaria o de mayor patogenicidad. La primera variante reportada fue la denominada inglesa (20I/501Y.V1), que en la nomenclatura PANGO (Phylogenetic Assignment of Named Global Outbreak Lineages) se identifica como B.1.1.7 y que presenta, entre otras, la mutación N501Y31. La mutación en la posición 501 altera la unión al receptor celular aumentando la transmisión del SARS-CoV-2. A la fecha, esto ha sido confirmado con diversos estudios usando estrategias que van desde el modelaje in silico hasta metaanálisis de datos.
Otra mutación que se ha encontrado de forma recurrente en diferentes partes del mundo es el cambio de ácido glutámico a lisina o a asparagina en la posición 484 (E484K o E484Q). Este cambio no afecta directamente la unión al receptor celular; los estudios recientes indican que es un sitio importante de reconocimiento de anticuerpos neutralizantes, y el surgimiento de virus con este cambio implicaría una evasión de la respuesta inmunitaria32. Otras mutaciones que afectarían la inmunogenicidad de la proteína S y que están surgiendo en diferentes partes del mundo son K417T, L452R, N439K y Y453F28. También se han detectado deleciones en la proteína S que afectan su estructura. Estas deleciones se han encontrado principalmente en dos posiciones: 69-70 (Δ69-70) y 144 (Δ144)31. En particular, se ha descrito que estas deleciones generan una disminución en la unión a los anticuerpos neutralizantes que son generados en pacientes tratados con plasma de pacientes convalecientes y de pacientes recuperados que presentan algún tipo de inmunodeficiencia33. El surgimiento de estas deleciones en estos grupos de pacientes podría sugerir que están siendo seleccionadas de forma positiva, y podría ser un mecanismo de resistencia a anticuerpos contra el SARS-CoV-230. Los sitios donde se encuentran estas deleciones se han denominado regiones de deleción recurrente, y aunque las sustituciones son relativamente poco frecuentes, podrían constituir un mecanismo que impulse una evolución rápida, lo que podría promover la deriva génica34.
Variantes virales
Actualmente, es posible clasificar al SARS-CoV-2 mediante la detección de mutaciones en su secuencia genética. A partir de las mutaciones fijadas en las poblaciones alrededor del mundo se pueden determinar sus variantes35. Una variante es un linaje que tiene un fenotipo demostrablemente diferente al de otras que circulan por el mundo, con dos o más mutaciones que confieren beneficios al virus, tales como antigenicidad, transmisibilidad o virulencia36. Las variantes se clasifican en variante de interés (VOI, variant of interest), variante de preocupación (VOC, variant of concern) y variante de gran consecuencia (VHC, variant of high consequence)36.
A finales del año 2020 se comenzaron a detectar diversas variantes del SARS-CoV-2 que circulan en diferentes poblaciones alrededor del mundo. Las principales VOC son las encontradas en Inglaterra (B.1.1.7 o alfa), Brasil (P1 o gamma), Sudáfrica (B.1.351 o beta y B.1.529 o gamma) e India (B.1.617 o delta)31,37–40; en la tabla 1 se muestran las mutaciones que presentan cada una de estas variantes. De forma importante se debe hacer notar que algunas de estas variantes comparten mutaciones, principalmente las mutaciones N501Y, E484K, L452R y P681H35. De acuerdo con esto, es de vital importancia conocer los efectos de la presencia de estas variantes en la transmisibilidad, la patogenicidad y la inmunogenicidad, así como en la efectividad de las vacunas que actualmente se están empleando.
Tabla 1. Impacto en la transmisión, la patogenicidad y la inmunogenicidad de las variantes del SARS-CoV-2
| Variante (mutaciones en Spike) | Mutaciones en otros genes | Transmisión | Patogenicidad | Inmunogenicidad |
|---|---|---|---|---|
| B.1.1.7/alfa (del 69-70, del 144, N501Y, P681H) | ORF1ab:T1001I ORF1ab:A1708D ORF1ab:I2230T del I1288-9 ORF8:Q27 ORF8:R52I ORF8:Y73C N:D3L N:S235F |
Aumento de 50% (A) | Potencial incremento en la gravedad, basado en datos de hospitalizaciones e índices de letalidad | Mínimo impacto en la neutralización por sueros de pacientes convalecientes y vacunados (B) |
| P1/beta (E484K, N501Y, P681H) | ORF1ab:S1188L ORF1ab:K1795Q del 11288:9 ORF3a:G174C ORF8:E92K N:P80R |
Sin datos | Sin datos | Reducción considerable de neutralización por sueros de pacientes convalecientes y vacunados (C) |
| B.1.1.351/gamma (E484K, N501Y) | E:P71L N:T205I ORF1a:K1655N |
Aumento de 50% (D) | Sin datos | Reducción considerable de neutralización por sueros de pacientes convalecientes y vacunados (C) |
| B.1.529/ómicron (30 mutaciones en S y 15 en RBD) | 25 mutaciones distribuidas a lo largo del genoma viral | Aumento de 50% (E) | Reducción de gravedad | Reducción considerable por neutralización de sueros de pacientes convalecientes y vacunados (E) |
| B.1.617.1/delta (T95I, L452R, E484K, P681R, | ORF1ab:T1567I, T3646A, P4715L ORF1ab:G5530C ORF1ab:M5753I ORF1ab:K6711R ORF1ab:S6713A ORF3a:S26L M:I82S, N:R03M, Q377Y, ORF7a:N43Y, V82A |
Sin datos | Sin datos | Reducción considerable de neutralización por sueros de pacientes convalecientes y vacunados (F) |
|
Variantes virales según la nomenclatura PANGO/OMS y sus principales mutaciones en la proteína Spike. |
||||
En resumen, se sabe que la variante alfa presentó mayor transmisibilidad, mientras que las variantes beta, gamma y delta, además de tener mayor capacidad de transmisión, escaparon a sueros de personas convalecientes. Además, se ha visto también que algunas vacunas, como la desarrollada por Astra-Zeneca, presentan menores niveles de protección contra estas variantes (Tabla 1). La última VOC apareció a finales de 2021 (ómicron) y presenta al menos 30 mutaciones en la proteína S y 15 en la RBD. Estas mutaciones le confieren una mayor afinidad por el receptor ACE2 y una mayor evasión de la respuesta inmunitaria estimulada por la vacunación y por infecciones por otras variantes del SARS-CoV-2. En Latinoamérica se detectaron nuevas variantes diferentes de la encontrada en Brasil. En Colombia, desde finales de 2019 se detectó una variante (B.1.1.621 o mu) que comparte varias mutaciones con otras VOC, como las mutaciones E484K, N501Y y P681Q41. A pesar de tener estos cambios, aún no es considerada como una VOI ni una VOC. Otra variante de interés ya aceptada por la Organización Mundial de la Salud (OMS) es la denominada lambda, que se detectó primero en Perú y presenta las mutaciones L452Q y F450S en S36. Por otro lado, en México, nuestro grupo de trabajo y otras instituciones del país detectamos desde finales del mes de diciembre de 2020 la presencia de un nuevo linaje (B.1.1.519), que incrementó su presencia durante los meses de enero a marzo de 202142. Este linaje presenta dos mutaciones importantes, T478K y P681H, por lo que se sugirió proponerla como una VOI42. Este linaje surgido a finales de 2020 en el centro de México presenta, además, otras mutaciones en otros genes virales: N:G204R, N:R203K, ORF1a_P959S, ORF1a_I3618V, ORF1a_T3255I y ORF1a_T4175I.
La VOC ómicron, que surgió a finales de 2021, ha permanecido desde entonces y se ha diversificado en diversos linajes y sublinajes, siendo los últimos en aparecer los derivados del sublinaje BA.2.86.1 o JN.1. La variante ómicron, originalmente denominada B.1.1.529, se diversificó en dos ramas en BA.1 y BA.2, y esta última a su vez se diversificó en BA.4 y BA.5 a principios de 2022. Estas ramas se diversificaron a lo largo del tiempo, siendo las predominantes la subvariantes BQ derivadas de la BA.2. Por otro lado, se produjo una recombinación en dos sublinajes de BA.2, BJ.1 y BM.1.1.1, y en abril de 2023 se detectó la subvariante XBB, siendo la XBB.1.5 la predominante durante varios meses en todo el mundo43. En agosto de 2023, la OMS designó a la subvariante BA.2.86 como una variante bajo monitoreo debido a que posee 33 mutaciones en S y 14 en RBD, comparado con su antecesora BA.2. A partir de esta subvariante surgió la JN.1, y a finales de 2024 se detectaron las variantes KP, XEC y LP, esta última dominante en 202544.
Vigilancia molecular mundial y en México
Lo que sabemos del SARS-CoV-2 desde el punto de vista molecular se debe al conocimiento de los genomas virales presentes en las muestras clínicas. Esto se realiza mediante secuenciación de nueva generación y métodos bioinformáticos. Con estas metodologías se ha podido conocer el agente etiológico causante de esta nueva enfermedad y ha sido posible conocer la evolución viral durante más de 1 año desde su aparición, así como el surgimiento de nuevas variantes que pueden afectar el curso de la enfermedad y la vacunación. En México, diferentes instituciones de salud, incluido el Instituto Nacional de Enfermedades Respiratorias Ismael Cosío Villegas, así como instituciones académicas, han realizado esta vigilancia molecular y hasta el mes de junio de 2021 se habían depositado en la base de datos GISAID más de 10,000 genomas completos de la mayoría de los Estados de la República. Estos datos son necesarios para conocer mejor la pandemia en nuestro país, así como para generar conocimiento que nos ayude a afrontar la emergencia sanitaria en los siguientes meses y años. Asimismo, este conocimiento nos puede ayudar también en el entendimiento de la evolución del virus y su adaptación al humano.
Vacunación y perspectivas de control de la enfermedad
La pandemia causada por el SARS-CoV-2 ha sido el motor para el desarrollo de nuevas estrategias de vacunación, como son las basadas en RNA, y el empleo de otras que comenzaban a utilizarse, como son los vectores de adenovirus. Además, se han empleado varias estrategias ya conocidas con otras vacunas, como el uso de péptidos, proteínas recombinantes y virus inactivados, entre otras (Fig. 5)45.
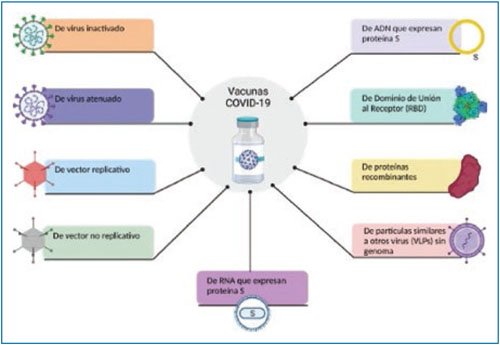
Figura 5. Repertorio de vacunas contra el SARS-CoV-2 desarrolladas empleando diversas estrategias; de las más exitosas, el empleo de RNA en nanopartículas por BioNTech-Pfizer y los vectores de adenovirus por el consorcio Oxford-AstraZeneca, el Instituto Gamaleya y la empresa Cansino.
Diversas instituciones académicas de diferentes países han realizado alianzas con grandes empresas farmacéuticas para el desarrollo masivo de vacunas. Actualmente, las vacunas más empleadas en todo el mundo son la desarrollada por BioNTech y Pfizer usando RNA, la de Oxford y AstraZeneca, la del Instituto Gamaleya ruso y la del Instituto de Beijing43, la Cansino usando vectores de adenovirus, y la vacuna inactivada de Sinovac. De estas, la vacuna que ofrece mayor protección es la de Pfizer, cercana al 90%, mientras que la de Cansino es la de menor protección (50-60%)46,47. La aparición de nuevas variantes ha sido una situación desafiante para la eficiencia de las vacunas. Las vacunas basadas en RNAm han incluido en sus nuevas formulaciones las variantes XBB.1.5 y JN.1 como actualización de la proteína S. Aunque estas nuevas vacunas estimulan anticuerpos neutralizantes contra las nuevas variantes, aún no existen estudios suficientes para conocer si existe una ventaja con respecto a la disminución de casos graves de la enfermedad48.
Ante la falta de fármacos antivirales de fácil acceso, actualmente la vacunación es la medida de control más efectiva, que a mediano y largo plazo dará como resultado la disminución de casos activos, de hospitalizaciones y de fallecimientos. La caracterización molecular del SARS-CoV-2 es primordial para monitorear la aparición de mutaciones en el virus que puedan afectar la efectividad de la vacuna o dar lugar a la aparición de variantes más patogénicas49.
Financiamiento
Ninguno.
Conflicto de intereses
Los autores declaran no tener conflicto de intereses.
Consideraciones éticas
Protección de personas y animales. Los autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad, consentimiento informado y aprobación ética. El estudio no involucra datos personales de pacientes ni requiere aprobación ética. No se aplican las guías SAGER.
Declaración sobre el uso de inteligencia artificial. Los autores declaran que no utilizaron ningún tipo de inteligencia artificial generativa para la redacción de este manuscrito.